
发表期刊:Environmental Pollution
影响因子:6.792
研究方法:m6A- meRIP-seq、RNA-seq、qPCR
文章链接:Global N6-methyladenosine profiling of cobalt-exposed cortex and human neuroblastoma H4 cells presents epitranscriptomics alterations in neurodegenerative disease-associated genes
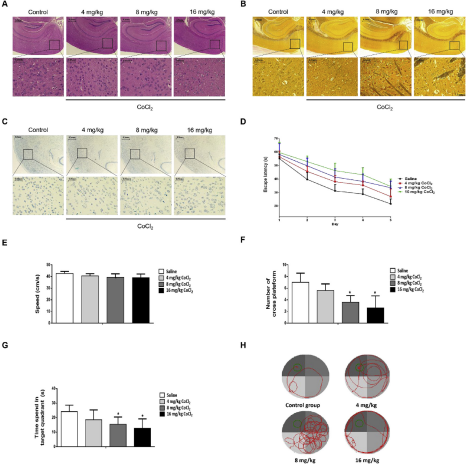
(1)CoCl2引起神经系统的病理和神经行为损伤
为了鉴定CoCl2会造成什么病理损伤,作者采用H&E染色法、Bielschowsky银染和Nissl染色法检测发现暴露于CoCl2后,皮质细胞数量显著减少,神经纤维缠结的数量增加,Nissl体减少。为进一步鉴定CoCl2对神经系统的影响,作者通过在小鼠中进行Morris water maze评估发现随着时间的增长,小鼠在CoCl2中逃跑的潜伏期逐渐缩短。此外,中、高剂量CoCl2组小鼠在目标象限停留的时间更少,通过平台的次数也明显低于对照组。根据到达平台所需的距离和到达次数间接反映小鼠的空间学习记忆能力,根据轨道图可知神经行为损伤的小鼠到达平台的距离较长,到达平台的次数较少。综上可知,长时间暴露于CoCl2中会导致空间学习和记忆障碍。
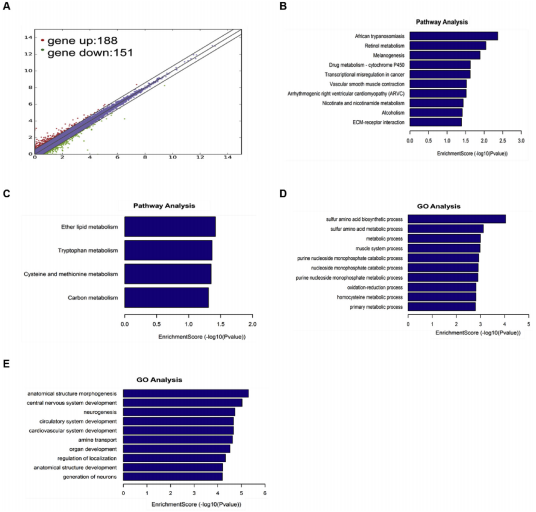
(2)GO和KEGG分析大脑皮层中受CoCl2影响的差异表达基因
通过RNA-seq发现在CoCl2中有339个基因表达水平发生了变化,其中188个基因表达上调,有151个基因表达下调。KEGG通路分析发现差异上调基因主要富集于睡眠、视黄醇代谢、药物代谢等方面;差异下调基因主要与代谢通路相关。GO分析表明,上调表达的基因在中枢神经系统发育、神经发生、氧结合等方面丰富;下调基因主要富集于多种代谢途径。
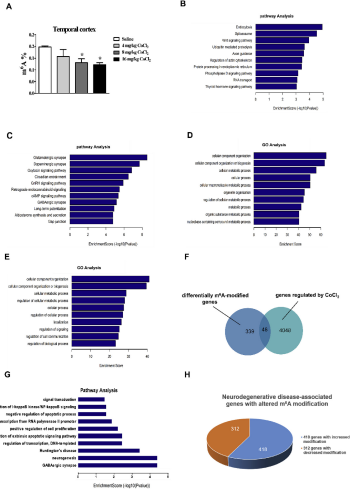
(3)CoCl2导致了大脑皮层基因m6A修饰的改变
m6A 比色法 鉴定m6A修饰总体水平发现,与对照组相比暴露于CoCl2的小鼠皮质内的总RNA的m6A修饰水平显著降低。使用m6A-MeRIP-seq显示,与对照组相比CoCl2处理组中有4048个基因的m6A修饰发生改变,其中2446个基因m6A修饰增加,1602个基因m6A修饰减少。GO和KEGG通路分析用于鉴定差异甲基化基因,高甲基化基因主要参与胞吞、剪接、信号转导、突触传递、RNA转运等通路而低甲基化基因主要富集于谷氨酸能、多巴胺能和GABA突触相关性。此外有48个基因既发生m6A修饰又受CoCl2调控,这些基因参与了神经元发育、细胞增殖和迁移途径。进一步观察发现CoCl2处理后有730个基因与神经退行性疾病相关,其中418个基因m6A修饰增加,312个基因的m6A修饰减少。
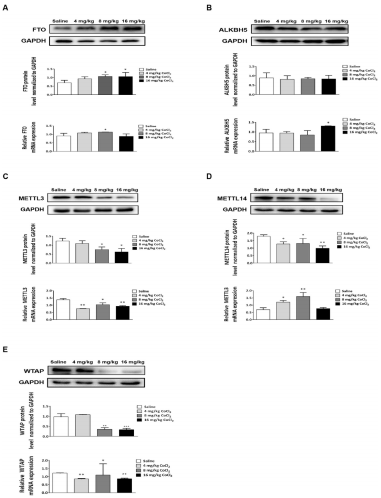
(4)CoCl2诱导大脑皮层中m6A修饰和基因表达变化机制
为了研究CoCl2处理如何改变m6A修饰并影响了基因表达。作者通过WB和qRT-PCR检测了去甲基酶(FTO和ALKBH5)和甲基转移酶(METTLL3、METTL14和WTAP)分别在处理组和对照组表达,结果发现FTO和ALKBH5蛋白和mRNA在处理组的表达水平增加,而METTL3、METTL14和WTAP蛋白和mRNA表达水平降低。这些发现表明,CoCl2调控m6A甲基转移酶和去甲基化酶的表达水平,从而改变了m6A修饰和基因表达。
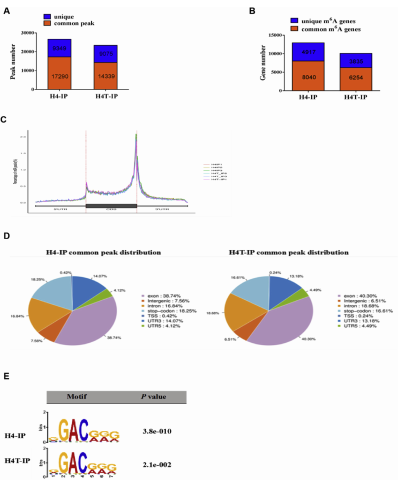
(5)H4细胞中m6A峰的差异分布
作者在H4细胞中进行了m6A-MeRIP-seq,首先分析了所有m6A的motif分布,结果显示对照组中有17290个的共有峰和9349个特异的m6A峰,在处理组中有14339个共有峰和9075独特的m6A峰。根据6个细胞样本中m6A峰的转录组分布,90%以上的峰位于基因区(5 ' -UTR、CDS、3 ' -UTR、内含子、停止密码子),不到20%的峰位于基因区(5 ' -UTR区和3 '-UTR区),60%以上的峰位于CDS区。同时,超过23%的富集m6A峰包含GGACG序列。此外,m6A-MeRIP-seq数据中与对照组相比,共有8752个基因甲基化,其中3835个基因发生高甲基化,而4917个基因发生低甲基化。
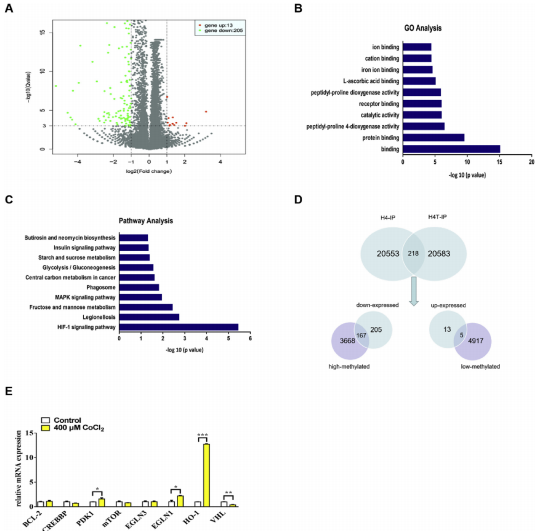
(6)GO和KEGG通路分析CoCl2处理H4细胞后表达变化的基因的富集情况
利用RNA-seq在H4细胞中寻找CoCl2调控的基因,有218个基因在CoCl2处理后出现,其中205个基因下调,13个基因上调。通过GO分析,差异基因主要富集于金属离子结合等方面;KEGG通路分析发现,差异表达基因在MAPK、HIF、肿瘤代谢以及凋亡、增殖和氧化相关通路中富集。进一步分析发现,有172个既发生了表达变化又有m6A修饰改变,167个高m6A修饰基因表达下调,5个低m6A修饰基因表达上调。对m6A修饰的氧化应激相关基因qRT-PCR分析结果显示PDK1、EGLN1、HO-1和VHL的表达在两组间有显著性差异。
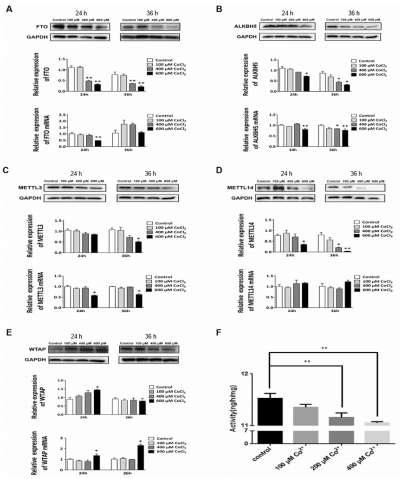
(7)CoCl2改变了m6A的甲基转移酶和去甲基化酶从而调节RNA m6A甲基化
对H4细胞进行不同浓度的CoCl2(100、400和600 mM)处理。用WB和qRT-PCR检测去甲基化酶(FTO和ALKBH5)和甲基转移酶(METTLL3, METTL14, WTAP)。结果证实CoCl2组中FTO、ALKBH5、METTL3、METTL14的表达水平下降,而WTAP蛋白表达水平升高。接下来,进一步验证CoCl2是否会抑制m6A去甲基酶活性,作者用m6A去甲基化酶激活/抑制测定试剂盒检测了CoCl2对m6A去甲基酶活性的影响。细胞暴露于不同的CoCl2 24h后,m6A去甲基化酶活性呈浓度依赖性下降。这些结果表明,CoCl2不仅调节m6A甲基化酶和去甲基化酶的表达,也降低了m6A去甲基化酶的活性,从而导致m6A甲基化改变。
文章通过m6A-meRIP-Seq结合全转录组测序技术(云序提供),通过对测序结果比对分析确认m6A甲基化修饰在CoCl2介导的神经毒性中起着重要作用,文中结合这两个测序结果取交集缩小差异范围,明确检测目标。另外MeRIP-qPCR结合WB实验(云序提供),验证CoCl2处理可以降低m6A修饰酶和去甲基化酶的表达以改变受m6A甲基化调控的基因表达,从而导致一系列的生物学不良反应。这些发现不仅有助于我们更好地了解疾病的病因,而且为重金属的治疗模式提供了新的靶点。
云序生物m6A修饰研究五大模块
01 m6A RNA修饰测序
m6A RNA修饰测序(m6A-seq)
对m6A RNA甲基化,目前流行的检测手段为m6A-Seq技术,适用于m6A RNA甲基化谱研究,快速筛选m6A RNA甲基化靶基因。云序可提供mRNA和多种非编码RNA的m6A测序:
- m6A 全转录组测序(涵盖mRNA,LncRNA,circRNA)
- m6A LncRNA测序(涵盖LncRNA和mRNA)
- m6A Pri-miRNA测序(涵盖Pri-miRNA和mRNA)
- m6A mRNA测序
- m6A miRNA测序
02 检测整体m6A RNA修饰水平
LC-MS/MS检测整体RNA修饰水平
准确高效,可以实现一次检测,9类修饰水平检测,一步到位。
比色法
快速检测m6A整体甲基化水平
m6A RNA修饰相关酶PCR芯片
寻找上游直接调控m6A RNA甲基化的甲基转移酶。
meRIP-qPCR
云序提供各类不同修饰的meRIP-qPCR服务,可针对mRNA,lncRNA,环状RNA等不同类型的RNA分子进行检测,低通量验证RNA修饰靶基因表达水平。
5.1 RIP-seq/qPCR
筛选或验证RNA修饰直接靶点,研究RNA修饰靶基因的调控机制。
5.2 RNA pull down -MS/WB
筛选或验证目标RNA互作基因或蛋白,研究相应的分子调控机制。
5.3 双荧光素酶实验
验证两基因互作,研究相应的分子调控机制。
优势一:发表10分以上文章多的m6A RNA甲基化测序服务平台。云序已累计支持客户发表36篇高水平文章,合计影响因子232分,是国内支持发文多、累计影响因子高的公司。
优势二:检测mRNA和各类非编码RNA(circRNA,lncRNA, Pri-miRNA等)。
优势三:提供m6A一站式服务:m6A整体水平检测、m6A测序、MeRIP-qPCR验证、RIP和RNA pull-down等。
优势四:率先研发超微量MeRIP测序技术,RNA量低至500ng起。
优势五: 国内RNA修饰测序平台,提供m6A、m5C、m1A、m7G、m3C、ac4C乙酰化和2'-O-甲基化测序。
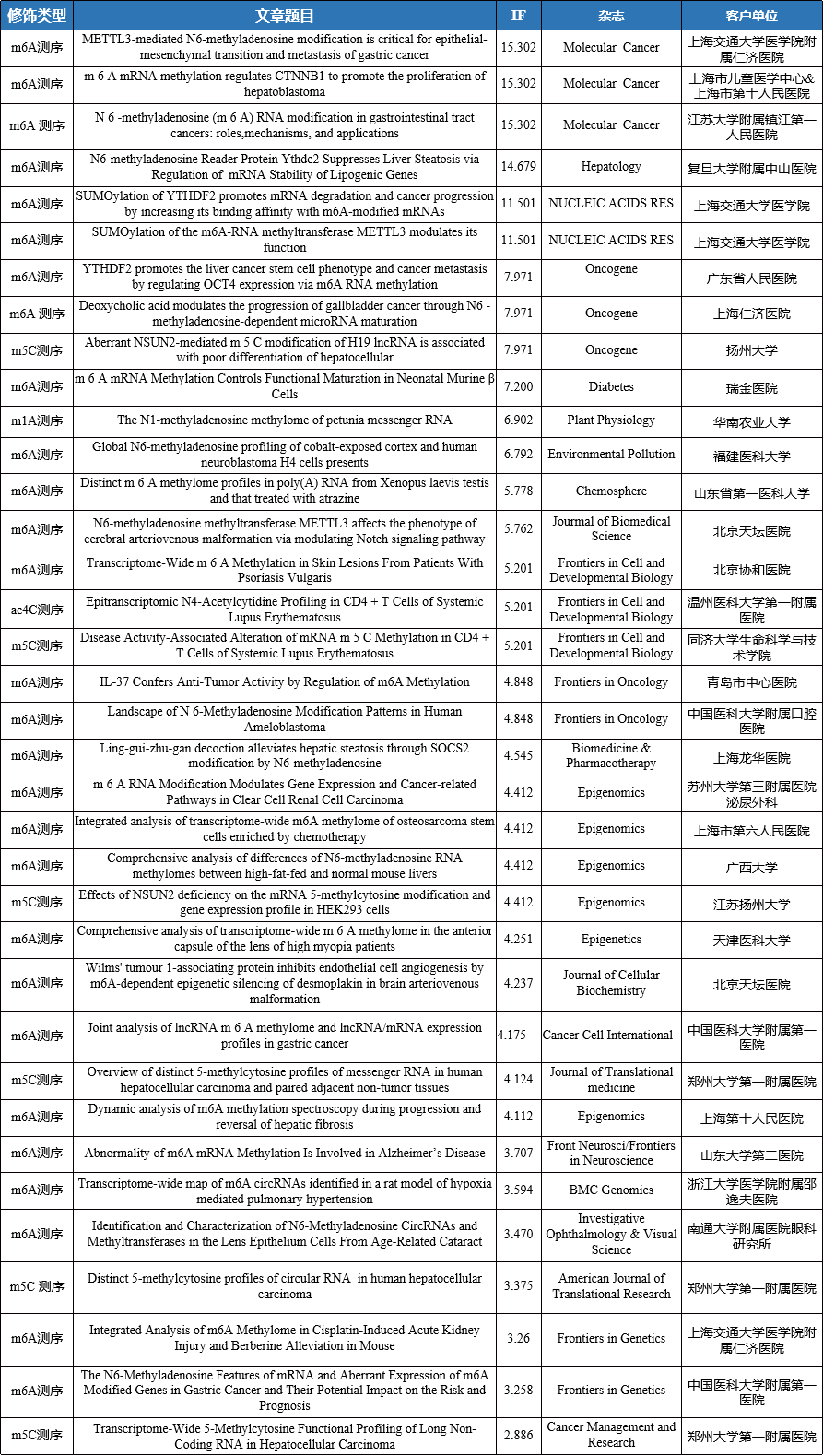
云序客户RNA修饰文章列表
相关产品
