荧光定量PCR是实验室中出镜率非常高的一种检测方法。该方法通过在PCR体系中添加荧光基团来记录DNA产物的累积情况,从而达到对PCR过程进行实时监控的目的,并且可以通过数据分析计算出起始模板量,这就是“荧光定量”中“定量”一词的来源。
荧光定量PCR实验因为灵敏度高所以经常是差之毫厘谬以千里,所以在实验过程中,我们需要注意诸多细节,谨慎操作。小伙伴们看到这里就要着急了,我怎样才能做好qPCR实验,拿到实验结果,发表高分文章,走上人生巅峰呢?
对于荧光定量PCR反应体系而言,cDNA模板的质量至关重要。cDNA模板的浓度、纯度、是否有一定程度的降解等等,都会影响荧光定量PCR的实验结果。绝大多数情况下,cDNA的质量和对应的RNA质量密切相关,高质量的RNA保证了高质量的cDNA。
RNA模块
RNA分离与纯化
基于有机试剂的一步萃取是一种非常有效的方法,能够从多种细胞和组织中分离纯化RNA。通常使用苯酚和异硫氰酸胍混合物(类似trizol)来破坏细胞并溶解细胞成分,同时异硫氰酸胍是一种离液盐,可保护核酸免受RNase的侵害以保证核酸的完整性。后加入氯仿并通过离心可将混合物分离成水相和有机相。在异硫氰酸胍存在下,RNA仅保留在水相中,而DNA和蛋白质则转移到有机相和中间相中,最后通过异丙醇沉淀从水相中回收RNA。
该过程相对来说较快,可以产生较多的RNA,但是其中需要使用有毒的化学物质,与其他RNA提取方法相比,可能导致更高的DNA残留。残留剩下的胍、苯酚或醇也会显著降低cDNA的合成效率。
使用硅胶膜或磁珠的方法时,生物样本会在异硫氰酸胍存在下进行裂解和均质(均质:由不同的成分互不相溶而产生均匀的混合物)。均质化后,将乙醇加入样品中,RNA会与硅胶膜或磁珠结合,并通过洗涤的方式去除杂质。该方法相较于有机萃取更省时,并且不需要苯酚。RNA产量可能没有有机萃取方式来的高,但是蛋白质、脂质、多糖、DNA等残留明显减少。当然,由于不完全洗涤,仍有可能发生胍和乙醇的残留,对cDNA合成效率产生一定的影响。
通常有机裂解和硅胶吸附相结合,可以做到样本充分裂解,RNA提取更加简便、快速和高纯度。
RNA提取三大纪律八项注意




RNA质量评估
常见评估RNA质量的方法有四种,分别是紫外光谱法(分光光度计测量)、核酸电泳、Qubit荧光测量和毛细管电泳分析法。
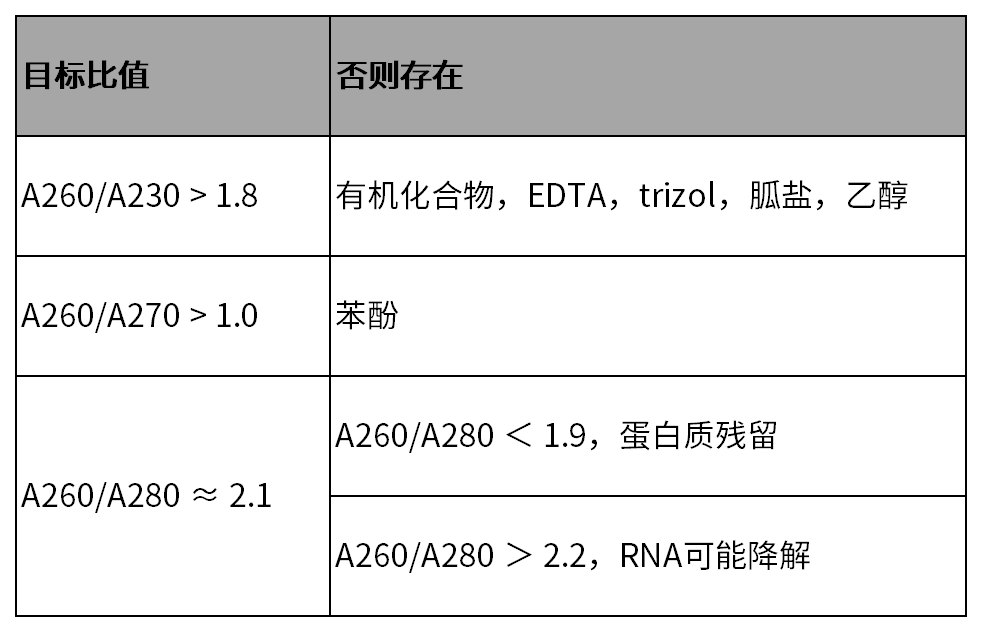
紫外光谱法是一种传统且简单的测量方法,大多数实验室均配备分光光度计。通常会检测三个值,分别是A230、A260和A280。
A230是盐类、多糖、有机溶剂等最高吸收峰的吸收波长,包括胍、EDTA、TritonTMX-100、Trizol、乙醇、多糖等最高吸收峰均接近230 nm。
A260是核酸最高吸收峰的吸收波长,由于核酸结构中的芳香碱基部分,包括嘧啶碱基(T、C、U)与嘌呤碱基(A、G)的最高吸收峰均在260 nm,因此A260值可用于核酸定量计算。
A280是蛋白和酚类物质最高吸收峰的吸收波长,蛋白质中色氨酸、苯丙氨酸、酪氨酸与胍氨酸的芳香氨基酸链与有机化合物中的芳香性苯酚的最高吸收峰均接近280 nm。
其中A260/A280>2.2表示RNA降解常为经验判断,并没有文章验证依据。判断RNA是否发生降解最好的办法是跑电泳进行鉴定。
紫外光谱法虽然高效方便,但其也存在一定的不足。紫外吸光度可能会受到以下几点的影响:
- 游离核苷酸的存在。吸光度不能区分核酸和游离核苷酸。并且游离核苷酸在 260 nm 处比核酸吸收更多,数值会受干扰,而非真实数据。
- 紫外吸光度测量无法区分同一样品中的RNA和DNA。
- 纯化核酸样品中通常存在的污染物(例如蛋白质)有助于紫外吸光度读数。
另一种常见的RNA质量评估方法是琼脂糖凝胶电泳。该方法可以判断RNA样本的降解情况、基因组残留和蛋白残留情况。
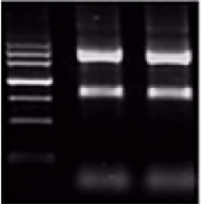
▲高质量的RNA电泳会有三条比较明显的条带,分别是28S,18S与5S核糖体RNA条带,并且28S与18S的条带亮度之比大体为2:1。当RNA出现降解时,条带会呈现弥散状
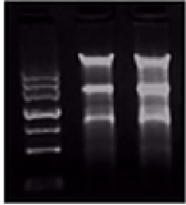
▲当基因组DNA有残留时,泳道上部会有明显的高分子DNA条带

▲当存在蛋白残留时,点样孔中会有比较亮的着色成分
RNA样本中DNA残留
当RNA样本中存在基因组DNA(gDNA)残留时,可能会在下游PCR实验或者下游荧光定量PCR实验中出现非特异扩增。在荧光定量PCR中直接的表现是Ct值明显偏小,且熔解曲线出现双峰。
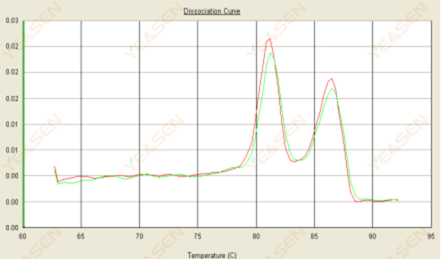
目前有三种方法可以避免RNA样本中gDNA的干扰。其中最有效的方法是做好引物设计,跨内含子且不在单个外显子模块中设计上下游引物。
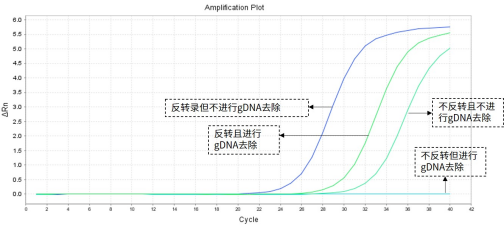
当引物设计不可避免或已完成了引物合成,则可以在RNA提取时选取带有gDNA去除功能的RNA提取试剂盒或在反转录获取cDNA时选用带有gDNA去除功能的反转录试剂盒。
cDNA模块
cDNA的合成
在正式进行cDNA合成之前,我们需要明确cDNA合成的模板是什么,是作用于RNA沉默和基因表达转录后调控的MicroRNA?还是涉及多个细胞过程的小型非编码RNA?还是将DNA信息转录为蛋白质产物氨基酸序列的mRNA?还是其他类型的RNA?不同的RNA合成cDNA所需要的引物会有所不同,应按需调整。
以RNA为模板合成第一链cDNA的过程为反转录。该步骤是反转定量中变化最大的一步,该步骤中可以使用随机引物、Oligo dT或序列特异性引物,引物选择在反转录效率、一致性和准确性上有着重要的作用。
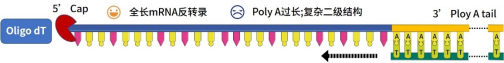
Oligo dT引物是常见反转录反应的首选,因为其对mRNA具有特异性,并且可以从同一cDNA库中分析许多不同的靶标。然而,由于其总在转录本RNA的3'末端启动反转录,二级结构的存在可能导致cDNA合成不完整。
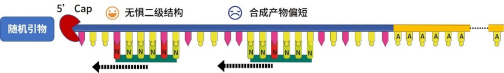
随机引物能够产生大量的cDNA,其在荧光定量 PCR 中具有最高的灵敏度。同时随机引物也是无poly A尾RNA的理想选择,例如原核生物RNA。随机引物在整个RNA转录本中退火,产生多个扩增位点,从而能够产生含有不同长度的cDNA。降解的RNA和复杂的二级结构对随机引物不会造成太大的影响。随机引物可以提高产量,但单独使用也会产生拷贝数被高估的风险。随机引物和Oligo dT引物的组合通常可以通过在同一反转录反应中结合两者的优点来以提高cDNA的质量。

基因特异性引物具有最大的特异性,并且是反转录引物选择中最为一致的。然而,这类引物没有Oligo dT和随机引物的灵活性,只产生靶基因产物所对应的cDNA。
cDNA浓度的测定
在正式测量cDNA浓度之前,可能需要了解一下,cDNA浓度是否需要测量?通常反转录后所获得的cDNA产物是一个混合体系,其中包含cDNA、未完全反转录的RNA、游离的核苷酸(dNTP)、残余的引物以及各类蛋白和盐离子等成分,会干扰cDNA的吸光值,从而影响判断。
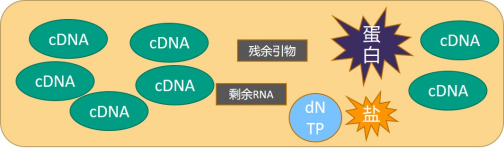
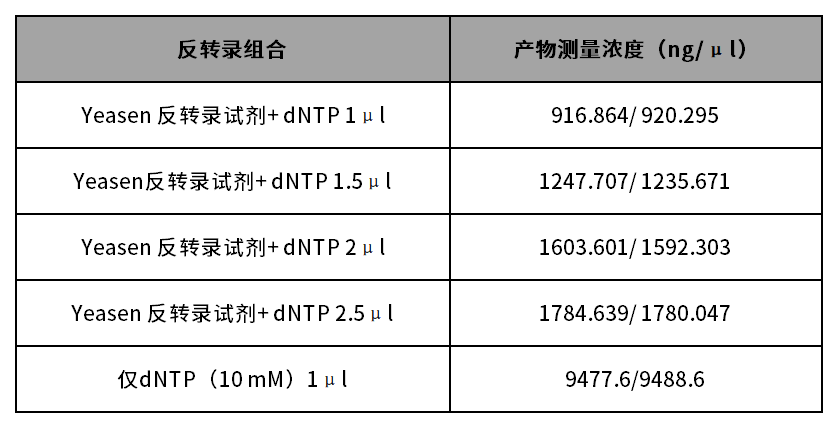
尤其是dNTP,该成分对于紫外光谱的影响较大。经实验验证,反转录体系中dNTP 投入量的多少会使cDNA浓度的测定结果产生较大差异,但最终Ct值几乎一致,qPCR检测结果△Ct<1。
▲Nanodrop定量结果统计表
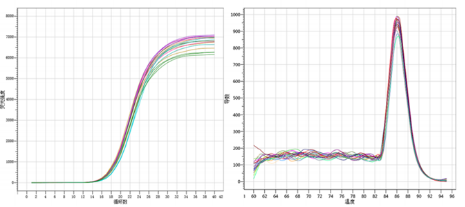
▲qPCR检测结果△Ct<1,且Ct值与 Nanodrop 定量结果无线性关系
cDNA浓度的测定受多因素影响,故可以选择不进行cDNA浓度的测定。另外同一批RNA浓度调整后,默认同一批次做的反转录效率是一致的,cDNA和RNA投入量呈现线性正相关,cDNA的浓度高低后续可通过荧光定量PCR反应中的内参数据来反映。故而在进行cDNA合成时,保证好高质量无降解的RNA,即可获取高质量的cDNA及准确的实验数据。
以上是对如何获取高质量cDNA的介绍,相信小伙伴们通过小翌的介绍,对如何获取高质量cDNA已经有一定的了解啦。关于如何做好qPCR实验,小翌会陆续在公众号里传授秘籍哒,敬请期待哦。

