随着高通量测序技术的发展,越来越多的非编码RNA(non-coding RNA,ncRNA)被注释,并且具有广泛的生物学功能,成为近年来的研究热点。本期小编为大家介绍两个最常见的研究RNA分子互作机制的实验技术:RIP(RNA结合蛋白免疫沉淀)和RNA pull down实验。研究与RNA、蛋白互作相关的文章,几乎都会涉及到这两个技术。为了让大家更好的理解运用这两个技术,小编将从技术原理,适用场景,云序客户案例解析三个方面进行剖析,告诉大家如何运用以及展示RIP、pull down技术结果!
RIP技术:是研究细胞内RNA与蛋白结合情况的技术。运用针对目标蛋白的抗体把相应的RNA-蛋白复合物沉淀下来,然后经过分离纯化就可以对结合在复合物上的RNA进行分析,结合的RNA可以通过定量PCR(RIP-PCR)或高通量测序(RIP-seq)方法来鉴定。RIP技术是研究转录后调控网络动态过程的有力工具。
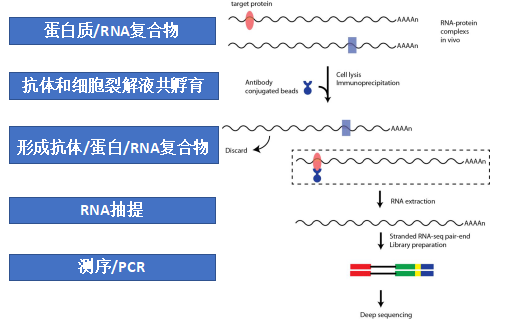
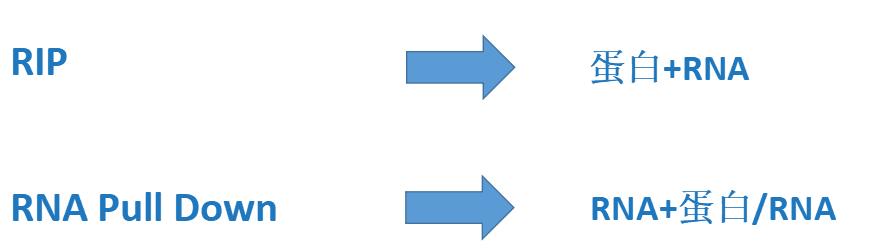
RIP技术:已知蛋白,探究与蛋白结合的RNA分子。此处RNA分子可以为mRNA以及非编码RNA(circRNA,lncRNA,miRNA,tRNA等。
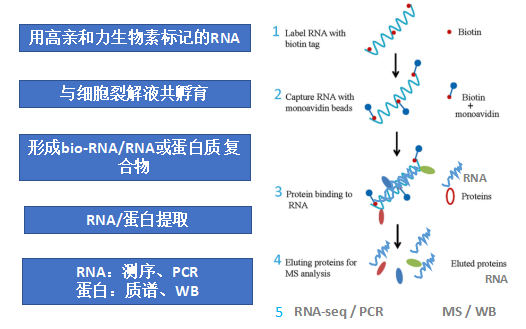
RNA pull down技术:已知RNA,探究与RNA结合的蛋白或RNA。此处RNA分子可以为mRNA以及非编码RNA(circRNA,lncRNA,miRNA,tRNA等)。
云序客户文章1:探究环状RNA的海绵机制
发表期刊:Cancer Gene Therapy
影响因子:4.681
实验方法:RIP-PCR、RNA pull down-PCR
实验对象:前列腺癌细胞
文献网址:https://sci-hub.shop/10.1038/s41417-019-0085-y
这篇文章主要讲述了circINTS4通过ceRNA机制促进膀胱癌恶化。作者是如何探究circINTS4的ceRNA机制的呢?其实跟lncRNA的ceRNA机制差不多。
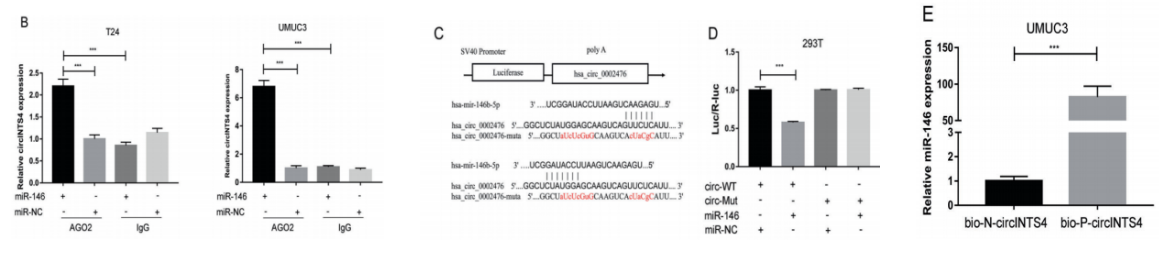
作者通过生信预测和前期文献报道,发现在circINTS4中含有miR-146b结合位点;那到底该如何探究circINTS4和miR-146b之间的关系呢?1)利用AGO2-RIP-PCR(云序提供该服务)证明,AGO2能够富集circINTS4和miR-146b(图B);2)通过双荧光素酶报告实验证明circINTS4与miR-146b存在结合位点(图C、D);3)作者通过circINTS4探针进行pull down PCR(云序提供该服务)实验,证明circINTS4 能够拉下miR-146b(图E)。
发表期刊:RNA Biology
影响因子:5.477
实验方法:CircRNA测序、RIP-PCR、RNA pull down-PCR、双荧光素酶报告实验
实验对象:鸡肝癌细胞
文献网址:https://sci-hub.shop/10.1080/15476286.2018.1564462
这篇文章主要研究circRNA Vav3通过吸附gga-miRNA-375促进鸡肝癌的EMT过程,也是运用了RIP-PCR(云序提供该服务)和RNA pull down-PCR(云序提供该服务)实验探究了circRNA Vav3能够吸附gga-miRNA-375的机制(图c-j)。
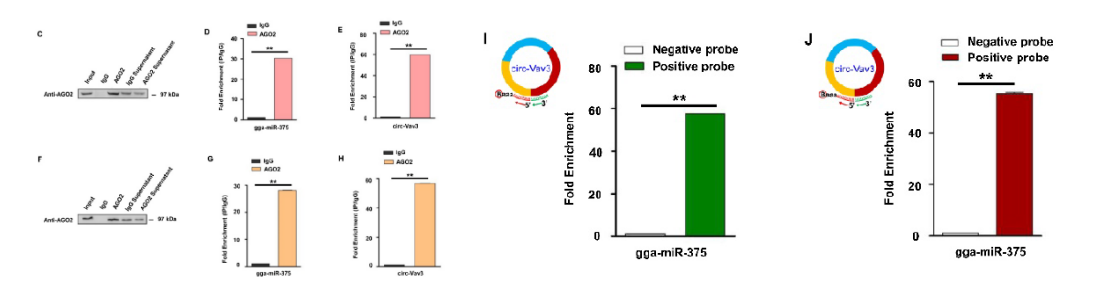
云序客户文章1:探究lncRNA的ceRNA机制
发表期刊:Cell Death & Differentiation
影响因子:8.086
实验方法:ChIP-qPCR、RIP-PCR、RNA pull down-PCR
实验对象:胃癌细胞
文献网址:https://sci-hub.shop/10.1038/s41418-018-0236-y
这篇文章主要讲述了被转录因子(KLF5和MYC)调控的LINC00346通过ceRNA机制促进胃癌的发生发展。那么在这篇文章中,主要运用什么手段进行研究的呢?作者首先通过生信预测,发现该LINC00346有多个miRNAs结合位点,然后通过LINC00346和miRNA之间的表达关系,将目标锁定在了miR-34a-5p上。那到底该如何证明,LINC00346能够吸附miR-34a-5p呢?1)通过LINC00346 Pull down PCR(云序提供该服务)进一步证明LINC00346能够拉下miR-34a(图C);2)通过双荧光素酶报告实验证明LINC00346与miR-34a-5p存在结合位点(图D);3)过表达miR-34a后进行AGO2-RIP-PCR(云序提供该服务)实验,证明过表达miR-34a后,LINC00346能够被明显富集(图E)。可以说作者把LINC00346的ceRNA机制这部分做的还是比较细致的。
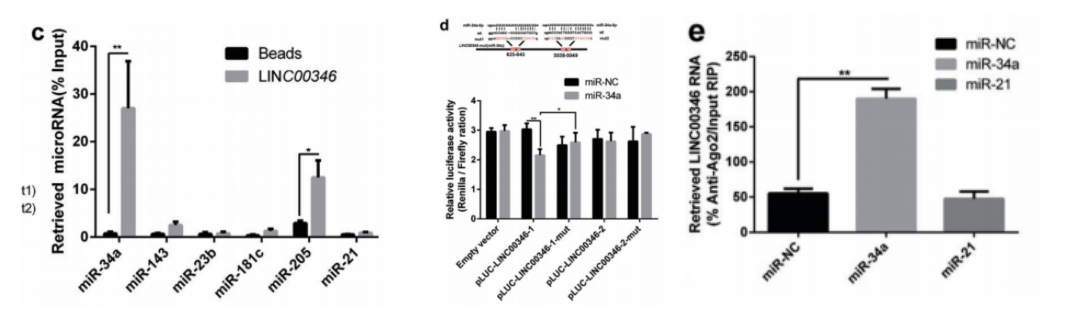
发表期刊:Oncogene
影响因子:6.634
实验方法:RNA pull down-PCR、双荧光素酶报告实验
实验对象:胃癌细胞
文献网址:https://sci-hub.shop/10.1038/s41388-019-0747-0
这篇文章主要讲述了MSC调节的lncRNA MACC1-AS1促进胃癌耐药。也是探究了lncRNA MACC1-AS1的ceRNA机制,那么小编为什么还是给大家介绍这篇文章呢?耐心往下看,您会发现,作者利用miRNA pull down实验,去探究lncRNA MACC1-AS作为ceRNA。也就是说用miRNA做pull down实验,仍然可以研究lncRNA吸附miRNA的海绵机制。作者利用生信预测和前期文献报道,miRNA-145-5和lncRNA MACC1-AS1存在结合位点;且发现lncRNA MACC1-AS1和miRNA-145-5p存在负性表达关系;1)作者首先通过双荧光素酶实验证明,miRNA-145-5p和lncRNA MACC1-AS1存在结合位点(图B)。2)为了进一步确定miRNA-145-5p和lncRNA MACC1-AS1之间的关系,作者利用miRNA-145-5p pull down-PCR(云序提供该服务)实验,证明miRNA-145-5p能够拉下lncRNA MACC1-AS1(图D)。
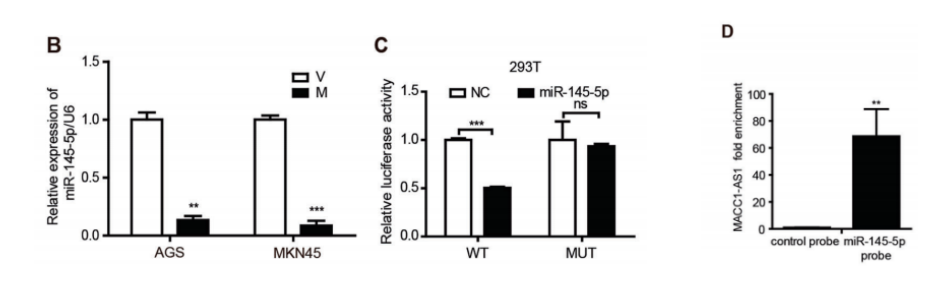
发表期刊:Oncogene
影响因子:6.634
实验方法:lncRNA测序、RIP-PCR、RNA pull down-PCR
实验对象:胃癌细胞
文献网址:https://sci-hub.shop/10.1038/s41388-018-0308-y
这篇文章主要讲述了lncRNA FOXD2-AS1通过结合蛋白(EZH2和LSD1)沉默EphB3调控胃癌的发生发展。那么是如何证明 FOXD2-AS1和EZH2/LSD1结合的呢?这篇文章作者通过生信预测,发现FOXD2-AS1能够与EZH2和LSD1结合;那么如何探究呢?作者利用EZH2或LSD1 RIR-PCR(云序提供该服务)实验,证明EZH2或LSD1确实能够结合FOXD2-AS1(图A);为了进一步验证,作者做了FOXD2-AS1 pull down WB(云序提供该服务)实验,证明FOXD2-AS1能够与EZH2和LSD1结合(图B)。
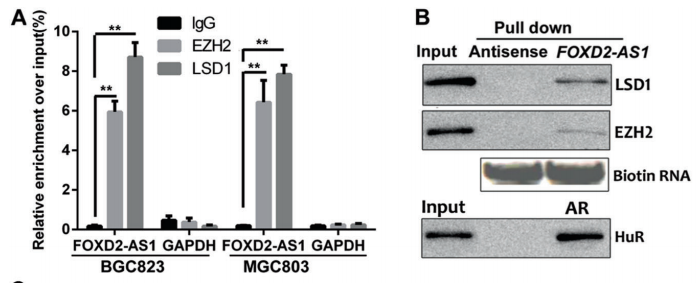
云序客户文章1:探究结合蛋白下游靶基因及机制
发表期刊:Genome Biology
影响因子:14.028
实验方法:RNA-seq、RIP-seq
实验对象:卵巢癌细胞
文献网址:https://sci-hub.shop/https://doi.org/10.1186/s13059-018-1412-6
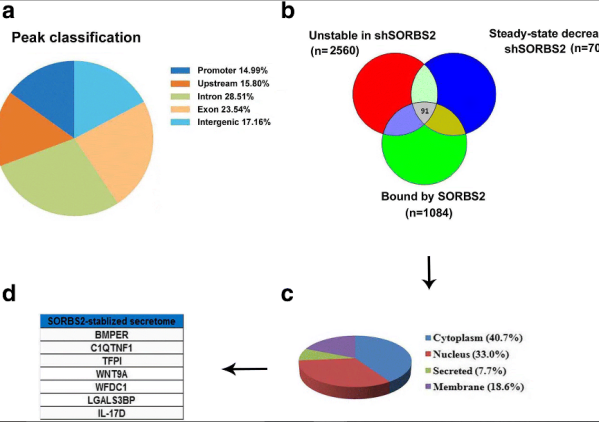
这篇文章巧用RIP测序解析RNA结合蛋白SORBS2在卵巢癌细胞转移中的作用机制。为了寻找SORBS2的潜在下游靶点,作者进行RIP-Seq(云序提供该服务) 技术,联合全转录组范围RNA稳定性实验以及RNA-seq(云序提供该服务),证明SORBS2可以与部分转录本结合并使他们更加稳定,并且从91个基因中仅锁定7个基因可以编码分泌蛋白,通过与SORBS2表达关系,最终找到了SORBS2的下游靶基因WFDC1和IL-17D(图a-d)。
发表期刊:The Journal of Clinical Investigation
影响因子:12.28
实验方法:miRNA测序、AGO2-RIP-qPCR
实验对象:血管平滑肌细胞
文献网址:https://sci-hub.shop/10.1172/jci124508
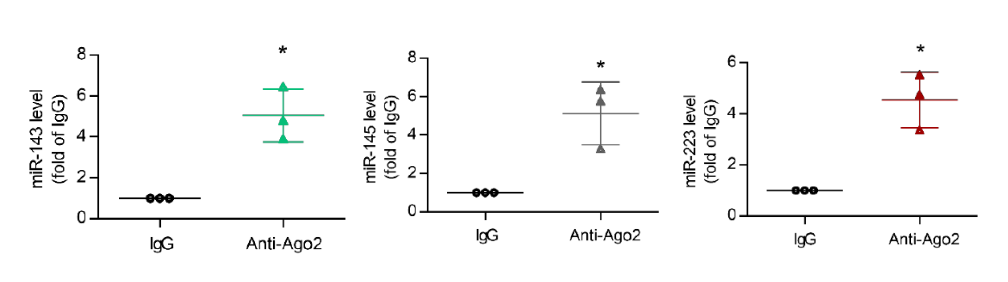
这篇文章主要讲述了血小板来源的miRNA能够促进主动脉损伤,为了识别血小板miRNA对血管平滑肌细胞的功能的影响,作者通过miRANA-seq(云序提供该服务)和AGO2-RIP-PRC(云序提供该服务)筛选出miR-223 和miR-143/145,并进一步对其下游靶基因进行探究。
发表期刊:MOLECULAR PLANT
影响因子:10.812
实验方法:tRNA测序、RIP测序、RIP-PCR、RNA pull down-PCR
实验对象:水稻
文献网址:https://sci-hub.shop/10.1016/j.molp.2019.04.012
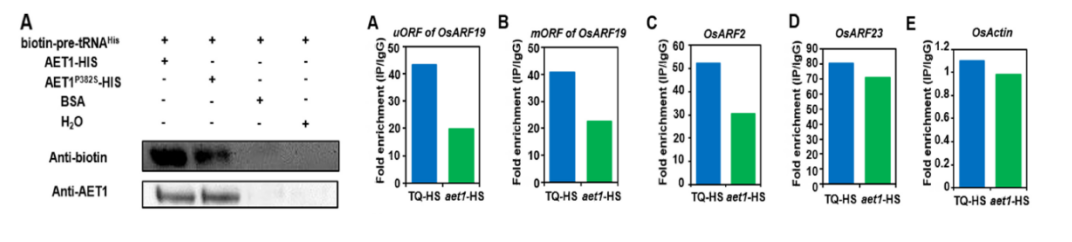
AET1是tRNA上鸟苷转移酶,它是水稻高温条件下正常生长所必需的。本文研究结果表明,tRNAHis鸟苷转移酶AET1在植物应对高温环境中发挥着重要作用。那么AET1是如何发挥功能的呢?作者通过RNA pull down WB(云序生物可提供)证明了AET1与RACK1A以及eIF3h结合,通过GST标签的RACK1A蛋白也能够富集eIF3H(图A)。AET1与RACK1A以及eIF3h的结合是否和mRNA相关?作者进一步利用RIP-seq(云序生物提供)快速明确哪些mRNA能够和蛋白AET1结合,分析结果显示AET1的功能丧失可以通过tRNA的表达分布的失衡从而影响蛋白的合成。比较有趣的是,热图提示高温条件下和植物生长素相关的基因在野生型和突变型当中有明显的改变,OsARF基因发生了明显富集,暗示AET1基因可能是通过改变植物生长素信号通路来应对高温产生应答反应。RIP-PCR(云序提供该服务)(图A-E)(云序生物可提供)结果显示OsARF19基因的上游开放阅读框(uORF)和下游主要开放阅读框(mORF)被明显富集。结果证明,是AET1,而不是RACK1A或者eIF3h广泛的结合OsARF mRNA来调控OsARF mRNA的翻译过程。
云序生物是业内少数的既做前端高通量测序筛选,同时实现一站式解决功能机制的科研服务公司。在各类RNA分子研究火热的现阶段,更多科研工作者想要在通过高通量筛选到目的基因后,解决RNA的分子机制问题,云序提供的RIP和RNA pull down技术正好解决分子机制的核心技术难点。此外RIP和RNA pull down技术同时也在当前最为火热的m6A RNA甲基化领域发挥了重要的应用,解决了m6A甲基化上游各类酶的机制研究,助力科研人员冲击高分文章。优质的测序服务,精心的差异挑选,准确的分子定位加上出色的pull down 、RIP一定能为您的科研工作增光添彩!
需要以上所有打包文献可以公众号留言or官网留言。
还在等什么呢?赶快致电云序,或与当地销售联系哦!
云序生物相关产品推荐
云序客户8.0分成果揭秘:还愁lncRNA功能机制如何研究吗?看这里!
云序客户13分文章,揭示糖尿病血小板来源miRNA参与海绵机制介导动脉损伤修复
Nature子刊|云序生物联合南京医科大发表重要研究成果:揭示lncRNA促进胃癌进展的新机制
年末惊喜不断!贺云序客户从表达谱至海绵机制完美进阶,教你轻松玩转环状RNA测序加机制研究!
上海云序生物科技有限公司
Shanghai Cloud-seq Biotech Co., Ltd.
地址:上海市松江区莘砖公路 518 号 20 号楼 3 楼

电话:021-64878766
传真:021-64878766
网址:www.cloud-seq.com.cn
邮箱:market@cloud-seq.com.cn
