一、听说最近 RNA甲基化很火,它是何方神圣?
1、高分文章频现
说起近来的科研热点, RNA甲基化修饰的相关研究可以说是当前整个生命科学领域最热门的方向之一,亮点文章频出,着实让人有些目不暇接。RNA甲基化的研究近3月发表的文章影响因子为10分以上的,就有高达 17 篇。

图: RNA甲基化近期高分文章一窥
2、Pubmed文章数屡攀新高
日益增多的发表文章、特别是高分文章说明,这个领域现在正在迅速成为大家关注的焦点。而就历年发表情况来看,RNA甲基化领域的增长势头也十分喜人,仅2018年1月已发表文献就达到了25篇。

图: RNA甲基化历年发文章数据
3、国自然立项数涨势喜人
纵向对比历年立项数,我们可以发现,RNA甲基化这两年的基金项目呈指数级增长的趋势:特别是从2015年的5项到2017年的25项,项目增长尤为显著。
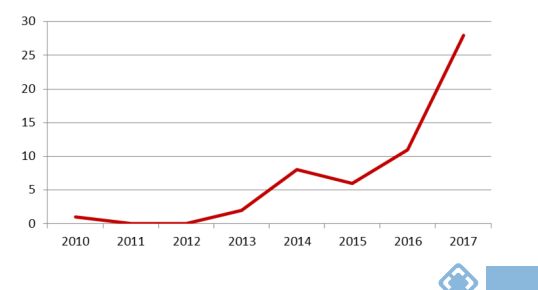
图: RNA甲基化历年国自然立项情况
由上图可知,2017年国家自然科学金获批的项目中, RNA甲基化研究相关的项目总数为25项。从获批的项目的研究内容来看,既有单纯鉴定RNA甲基化谱的课题,也有从RNA甲基化修饰酶入手的课题,以及针对RNA甲基化的功能机制展开的课题。这表明RNA甲基化的研究还是处于一个基础筛选与功能机制齐飞,比较多样化的状态。值得一提的是,也有课题将RNA甲基化修饰与miRNA以及外泌体分泌联系起来,以筛选/鉴定肿瘤分子标志物,这也是一个值得尝试的RNA甲基化重要方向。
咱们举例来看一下2017年的具体立项情况,我们也可以看出,除了鉴定RNA甲基化相关酶的切入点,RNA甲基化课题涵盖范围极其广泛,包括了:疾病发生发展、水稻生殖发育、果蝇性别决定等诸多重要领域。

图: RNA甲基化修饰2017国自然立项列举
二、听说有3种RNA甲基化修饰大热,他们是谁?
RNA甲基化修饰类型很多,目前最热门的有三种,分别是:m6A RNA甲基化,m5C RNA甲基化、m1A RNA甲基化。
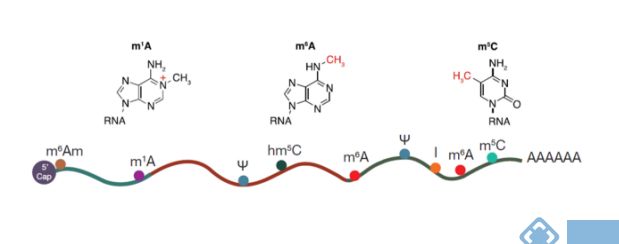
图: 3种热门RNA甲基化修饰
1、m6A RNA甲基化是最常见、最丰富的真核生物mRNA转录后修饰,现在的研究主要是围绕各修饰的关键酶展开。何川教授在2017年1月的Nature Review(IF=46.602)上总结过m6A的相关修饰,由甲基转移酶复合体(METTL3、METTL14和WTAP组成)、去甲基酶(FTO和ALKBH5)以及相应的阅读器(YTHDF1/2/3,YTHDC1)协同调控。相关功能的研究证明了m6A修饰是动态可逆的,而且具有种类多而又广泛的重要生物学意义。已发现的m6A RNA甲基化功能有很多,比如它可以促进环状RNA翻译(2017.3 Cell Res, IF 14.812),或者通过促进mRNA降解来调控癌症干细胞的分化(2016.4 Cell Host Microbe, IF 9.661),还能调控T细胞分化及免疫稳态(2017.8 Nature. IF 40.137)。
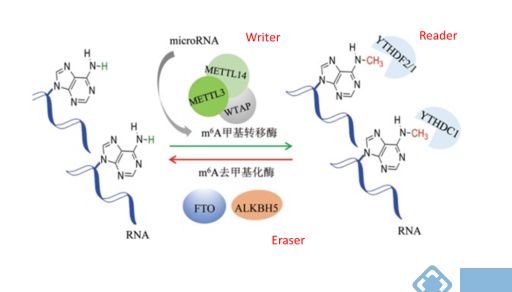
图: m6A RNA甲基化动态可逆修饰
2、m5C RNA甲基化 是今年来发现一类在tRNA及rRNA高丰度存在的甲基化修饰。利用高通量测序手段验证了非编码RNA以及部分mRNA中m5C广泛存在,但是不同物种、不同组织中m5C修饰分布图谱尚没有系统性报道。对于m5C来说,也有一系列的相关修饰酶(如下图),m5C RNA甲基化功能,目前发现他可以调控mRNA出核(2017 Cell Res, IF 14.812),或者调控干细胞蛋白质合成(2017 Nature, IF 40.137)。
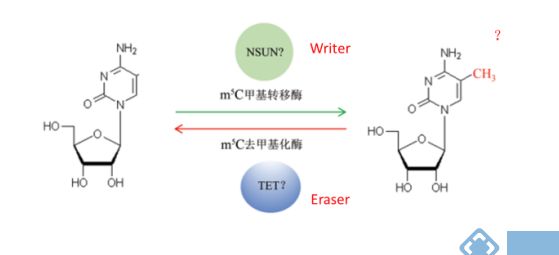
图: m5C RNA甲基化动态可逆修饰
3、m1A RNA甲基化 是一个新进入大家的视野的RNA甲基化修饰类型。最近发表在Molecular Cell (IF:14.714)上的一篇关于m1A RNA甲基化的文章介绍到这是一类新型RNA修饰,它普遍存在于非编码RNA和mRNA中。m1A RNA甲基化功能,目前发现tRNA上发生多种m1A修饰(2017.2 Nucleic Acids Research, IF 9.28),或者参与调控翻译起始和延伸(2016 Cell,IF 30.41)。总之,RNA甲基化也是调控基因表达的一种重要途径。
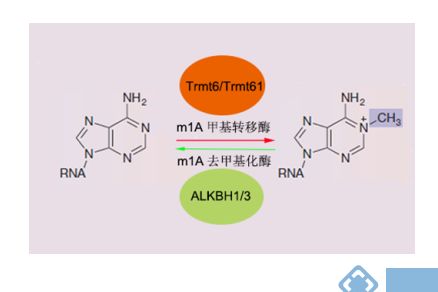
图:m1A RNA甲基化动态可逆修饰
三、RNA甲基化相关的酶有哪些?
RNA甲基化修饰相关蛋白包含特定甲基化修饰持有的甲基转移酶(Writer)、去甲基酶(Reader)以及相应的RNA甲基化识别蛋白(Reader)协同调控,以维持特定类型甲基化的动态修饰。许多高分文章都是针对甲基化修饰蛋白来进行功能机制研究。那么至今为止,不同的甲基化修饰的关键修饰蛋白都有哪些呢?经过翻阅文献查找,小编帮大家总结了一下目前文献报导的相关修饰蛋白,方便大家参考:
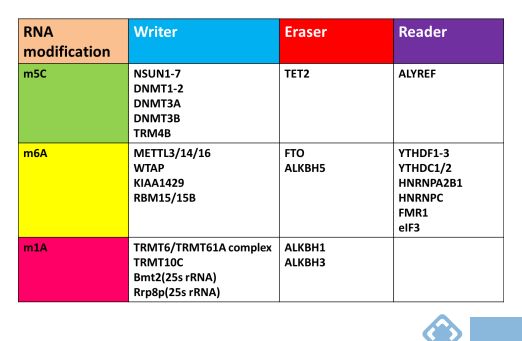
图: RNA甲基化修饰相关酶整理
四、 RNA甲基化这么火,它的研究思路是怎样的呢?
RNA甲基化目前的研究思路由浅及深,也是有两大类:1、RNA甲基化谱2、功能机制研究。那么且让咱们分门别类、细细道来!
1、RNA甲基化谱
由于RNA甲基化领域非常新,绝大部分物种、组织和疾病中的RNA甲基化分布图谱尚未报道。对于想要短平快的发表文章的老师来说,RNA甲基化谱分析是非常合适的。
一篇RNA甲基化谱的文章是怎么炼成的?研究伊始,我们要确定好研究目标:物种,疾病模型,样品类型、尽可能详尽的样品信息,接下来,通过RNA甲基化高通量地筛选RNA甲基化区域或位点,并进行深入的生物信息挖掘,例如:甲基化位点统计、基因组分布、甲基化基因功能进行预测(GO、Pathway分析)等等。
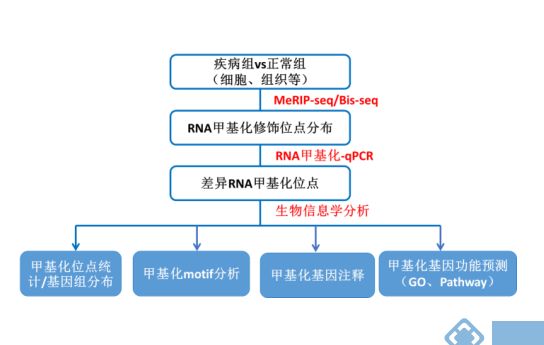
图: RNA甲基化表达谱思路图
咱们就以一篇影响因子11分的典型的RNA甲基化谱文章为例,来了解一下吧。这篇文章发表在Genome Biology上,展示了小鼠胚胎干细胞及大脑中的m5C RNA修饰整体情况。
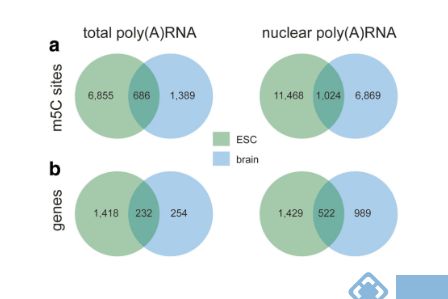
图: m5C RNA甲基化位点统计
首先通过 Bis-seq技术对不同样本中总poly(A)RNA和核poly(A)RNA的 m5C位点共分析和特异性分析。

图: qPCR验证m5C RNA甲基化测序结果
随后用qPCR验证m5C修饰位点,从图上可以看到16个验证的转录本中13个都显著富集。接着对m5C RNA甲基化测序的结果进行分析,发现大多数ESC总poly(A)RNA中特异的m5C甲基化修饰是由于甲基化程度的差异造成的,而与由不同样本间基因差异表达无关。并对m5C修饰的RNA对应基因进行功能预测,显示小鼠脑组织及小鼠胚胎干细胞间共有和特有的前十条GO条目。
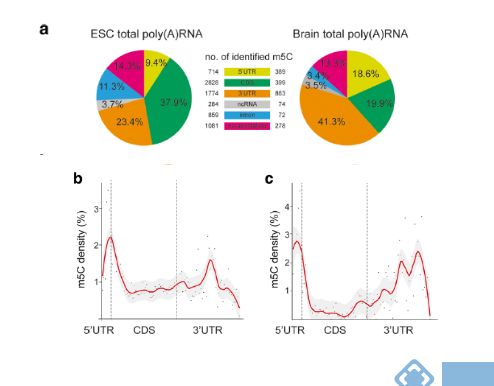
图:不同样本的 m5C位点分布规律
进一步的数据分析显示,不同样本的总poly(A)RNA中m5C位点一致的分布规律:小鼠胚胎干细胞中m5C修饰位点优先分布于mRNA其实密码子附近,在小鼠脑组织中则倾向于分布在3’UTR区。
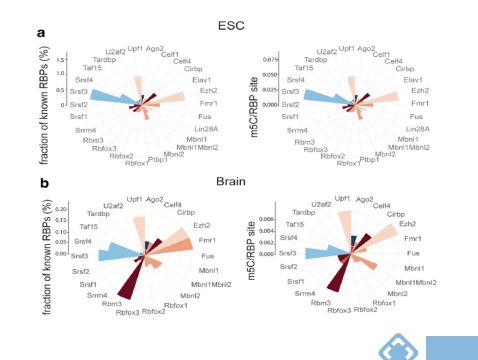
图: CLIPdb : m5C位点与RBPs结合位点的共分析
通过与CLIPdb的数据联合分析发现,小鼠胚胎干细胞中大约29%的poly(A)m5C RNA修饰位点与对应RBP(RNA结合蛋白)结合位点重合,这一百分数在小鼠脑组织中大约为11%,显示出与RBP结合并在特定转录本中发挥生物学功能的潜力。
到这里这样一篇m5C RNA修饰表达谱的文章就结束了,可以看出几乎没有涉及到实验操作,仅仅是针对m5C RNA甲基化测序的结果进行详尽的展示和分析。亮点是还联合CLIPdp的数据预测了一下m5C RNA甲基化位点作为RBP靶点的潜力。值得我们借鉴。
2、功能机制研究
看完了表达谱,那么RNA甲基化的功能机制要如何研究?咱们就以一篇影响因子27分的Cancer Cell文章为例,来了解一下吧。这是一篇来自于美国安德森癌症中心黄素云课题组与芝加哥大学何川课题组合作的课题,揭示了m6A RNA甲基化在神经胶质瘤中的作用。
首先放一下技术路线图。
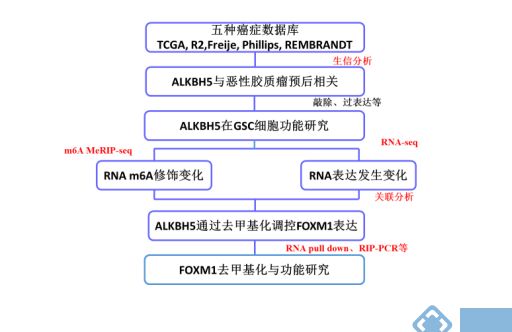
图:展示的文章功能集中技术路线图
TCGA数据库显示GSC中m6A去甲基化酶ALKBH5的表达水平明显升高。对不同病人的预后检测,生存曲线发现ALKBH5高表达的病人预后更差。
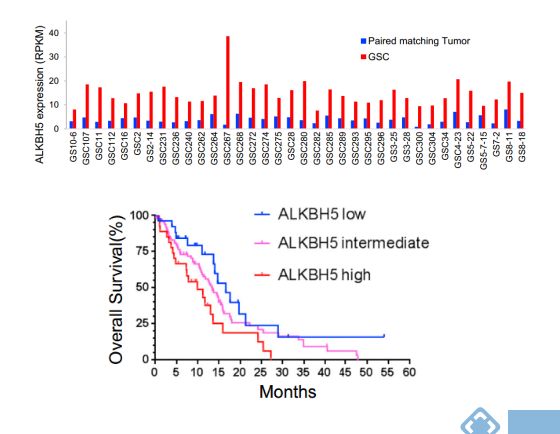
图:ALKBH5与较差的临床预后相关
作者通过MeRIP-Seq技术鉴定m6A RNA甲基化修饰图谱,联合表达谱检测差异表达>2倍的基因。最终筛选到与细胞周期调控相关的ALKBH5靶基因-FOXM1,其在GSCs的自我修复和肿瘤形成中起到关键作用。
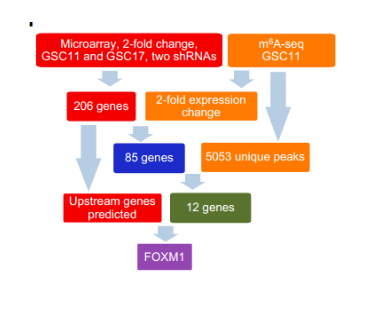
图: ALKBH5作用靶基因——FOXM1筛选流程图
通过MeRIP qPCR发现敲低ALKBH5可增加FOXM1前体mRNA的m6A甲基化水平。这些结果表明ALKBH5主要通过去甲基化活性影响FOXM1的表达。
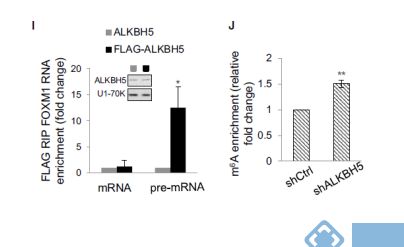
图:敲低ALKBH5上调FOXM1的前体mRNA表达及m6A RNA修饰
通过RIP、RNA pull down实验表明FOXM1-AS可与ALKBH5和FOXM1前体mRNA直接结合。
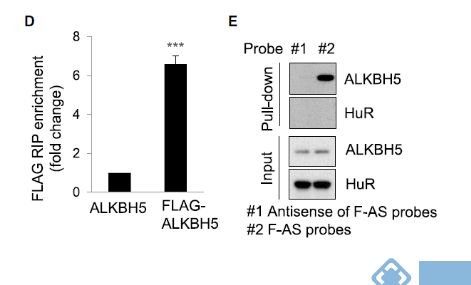
图:RIP和RNA Pull down实验证明ALKBH5和FOXM1-AS 直接结合
最后,作者进一步研究了FOXM1-AS对恶性胶质瘤细胞生物学功能的影响,结果表明FOXM1-AS可正调控FOXM1的表达,并可维持恶性胶质瘤细胞性能。而外源性的FOXM1可显著逆转敲低ALKBH5引起的抑制作用。
1、云序生物MeRIP-Seq
适用范围:m6A RNA甲基化﹑m5C RNA甲基化﹑m1A RNA甲基化;
技术原理:用特异性抗体富集甲基化RNA后再对其进行高通量测序检测;
目前对RNA甲基化最流行的检测手段为MeRIP-Seq技术,该技术将甲基化RNA免疫共沉淀和RNA测序技术组合起来,高精度地检测全转录组范围内的RNA甲基化修饰。具体原理如下:将针对特定RNA甲基化修饰的抗体与RNA样品共孵育(IP),抓取有甲基化修饰的RNA片段进行测序;同时需要平行测序一个对照(Input)样本,用于消除非特异性抓取甲基化片段的背景。对比免疫共沉淀样本(IP)和对照样本(Input)中的序列片段,将RNA甲基化修饰位区域定位到转录组上,并计算样本中RNA甲基化程度。云序生物具有全面的m6A﹑m5C﹑m1A甲基化MeRIP-Seq产品线,能够对circRNA,LncRNA,mRNA中的各类RNA甲基化修饰进行全面的检测。
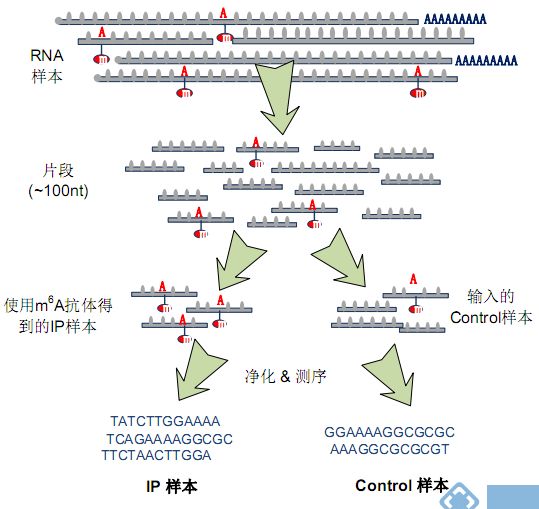
图:云序生物MeRIP-seq实验流程
2、云序生物Bis-Seq
适用范围:m5C RNA甲基化;
技术原理:对重亚硫酸盐转化后的RNA进行高通量测序检测;
针对m5C RNA甲基化的修饰还有一种单碱基分辨的检测手段:用重亚硫酸盐(Bisulfite)处理样品。重亚硫酸盐处理能够将基因组中未发生甲基化的C碱基转换成U,进行PCR扩增后变成T,这样反转录的产物就会出现单点突变,结合高通量测序技术,通过与不处理组进行序列比对,就可以很容易的找出m5C修饰的位点。云序生物具有成熟的Bis-seq实验平台以及丰富的RNA 甲基化测序经验,可以满足客户对不同样本、不同类型RNA 甲基化测序的各类需求。
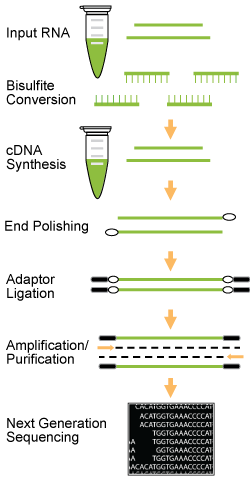
图:云序生物 m5C Bis-Seq 实验流程
3、云序生物RIP 和 RNA Pull down
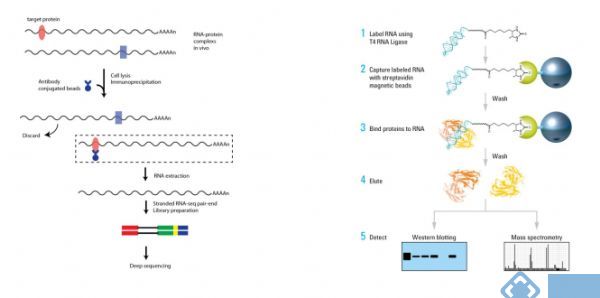
当测序、筛选和验证工作结束后,想要发表高分文章,那么功能机制实验的进行往往还少不了两个好帮手:RIP和RNA pull down实验,主要用来证实候选靶基因与甲基化相关蛋白的直接结合,是解析RNA甲基化调控机制的利器。就像上面举例的Cancer Cell一文里,作者通过 RIP、RNA pull down实验来证明受RNA甲基化调控的靶基因可与去甲基化酶ALKBH5直接结合。
那么RIP和RNA pull down是什么呢?
①RIP(RNA Immunoprecipitation)是RNA免疫共沉淀技术. 利用抗体特异性结合目的蛋白,进而检测与该蛋白结合的RNA分子,用于检测特定蛋白质与RNA的相互作用。RIP-Seq结合RIP技术和高通量测序,能够高通量地检测与特定蛋白结合的RNA,从而解析全转录组范围内的目标蛋白-RNA相互作用网络。RIP-qPCR则可以检测特定蛋白上结合的RNA分子。例如可以通过RIP-qPCR检测甲基化修饰相关酶是否与某RNA分子结合。
②RNA Pull down是利用特定探针把目的RNA拉下来,进而检测与之结合的RNA或蛋白质。用于检测特定RNA与蛋白质/RNA的相互作用。RNA Pull down后接Western Blot可以检测特定的RNA是否与特定的RNA结合蛋白相互作用。RNA Pull down后接质谱实验则可以解析蛋白质组范围内的所有蛋白信息。
图:云序生物RIP-seq及RNA Pull down实验流程
五、RNA甲基化项目国自然申请建议
1、RNA甲基化研究目前处于上升期,可以尝试设计特定细胞、组织类型表达谱的研究内