绝对定量测序热度持高,但是,为什么要做绝对定量测序呢?上期为大家解读了miRNA绝对定量测序的实验原因和实验解决策略(点此查看),今日小编为大家友情解读miRNA绝对定量测序的数据分析,话不多说,进入正题:
前面几篇软文已经提到,之所以要做绝对定量测序(转录组测序or miRNA测序),实验原因在于测序前文库构建过程,不管是转录组文库构建还是miRNA文库构建,均有一步PCR扩增的过程,而此过程由于PCR本身原因产生PCR Duplication,从而使得表达量失真,因此需要通过UMI技术,在后续分析过程消除PCR Duplication产生的测序数据,尽可能还原基因/miRNA表达的真实性。
有的看官可能会讲,即使不通过UMI技术,也可以在后续分析过程实现PCR Duplication的消除!是的,在高通量测序数据分析阶段,确实会做PCR Duplication去除,此时去除的理论依据是在所有相同的reads中只保留一条read,同时假设这些相同reads来自于由同一条cDNA PCR产生的相同扩增产物。具体的做法是通过mapping coordinates方法实现(reads比对到同一个基因组位点,会被认为是相同的reads)。但是这种方法有它的弊端:
1、高通量测序中,来自不同cDNA但是序列一致的reads出现的概率增大,使用mapping coordinates方法或人为丢失阳性数据;
2、对于基因组比较小,或者是基因组子集测序(例如miRNA测序,编码miRNA的区间只占基因组的很小一部分),此种去mapping coordinates导致的后果会更严重,因为序列越短,不同cDNA来源产生相同reads的概率越高;
3、具有相同序列的miRNA成熟体可由不同前体加工,对应不同的基因组位点,因此使用mapping coordinates方法会忽略掉此信息。
总结一下,常规mapping coordinates方法会导致真实的数据被错误的去除,造成数据的失真。所以,我们需要UMI技术克服这种问题。下面我们通过一些数据来展示UMI技术和mapping coordinates方法技术相比,在转录组测序和miRNA测序中去除PCR Duplication的优势。
表1:转录组测序两种PCR Duplication方法比较
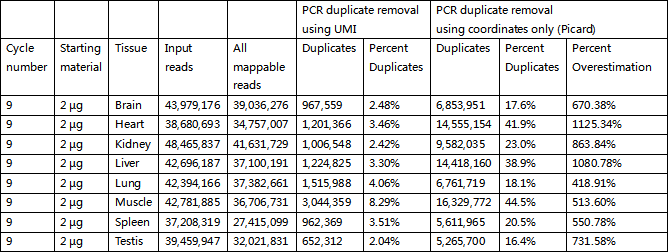
表2:miRNA测序两种PCR Duplication方法比较
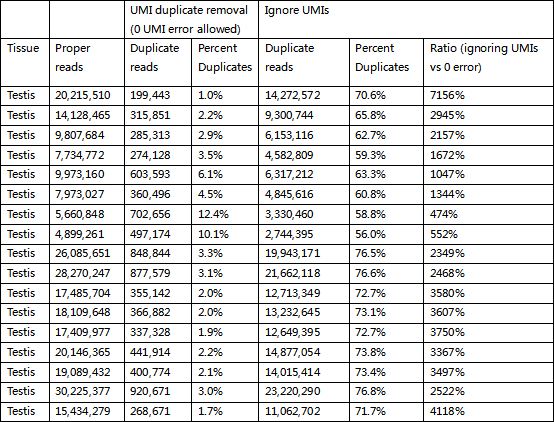
从两个表可以看出,转录组测序中使用mapping coordinates法,16.4–44.5%的reads被认定为PCR Duplication,而UMI方法只有1.89–10.67%被认为是PCR Duplication,高估率达到几百上千倍。而在miRNA测序中,mapping coordinates法认定的PCR Duplication数据极为夸张,达到了56.0–76.8%,实际上使用UMI方法分析得到的PCR Duplication比率只有1.05–13.6%,二者相比最低相差也达到千倍。
